Ecologia Numérica
Aula 3 - Medidas de diversidade biológica
Felipe Melo
Laboratório de Ecologia Aplicada - UFPE
2022-12-27
1 / 25
Diversidade Taxonômica

2 / 25
Precisamos saber:
3 / 25
Precisamos saber:
O que é diversidade?
3 / 25
Precisamos saber:
O que é diversidade?
Quais as métricas mais usadas?
3 / 25
Precisamos saber:
O que é diversidade?
Quais as métricas mais usadas?
Quais são os prós e contras?
3 / 25
4 / 25
5 / 25
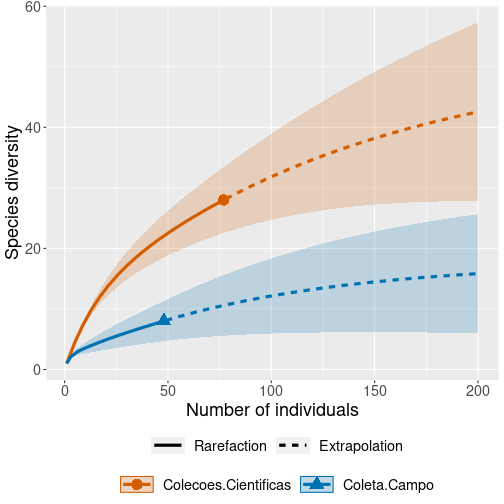
Será que amostramos todas as espécies?
6 / 25

7 / 25

8 / 25

9 / 25

10 / 25
11 / 25
12 / 25
13 / 25
O método mais atual
14 / 25
Exemplo de rarefação - Morcegos em 3 localidades
library(iNEXT)library(devtools)#devtools::install_github("paternogbc/ecodados")library(ecodados)dados_rarefacao <- rarefacao_morcegosresultados_morcegos <- iNEXT(dados_rarefacao, q = 0, datatype = "abundance", endpoint = 800)# q refere-se a família *Hill-numbers* (Hill 1973) onde 0 = riqueza de espécies, 1 = diversidade de Shannon, e 2 = diversidade de Simpson.# Veja mais detalhes sobre os números de Hill no Capítulo 7 onde tratamos de extrapolações.# datatype refere-se ao tipo de dados que você vai analisar (e.g. abudância, incidência).# endpoint refere-se ao valor de referência que você determina para a extrapolação.ggiNEXT(resultados_morcegos, type = 1)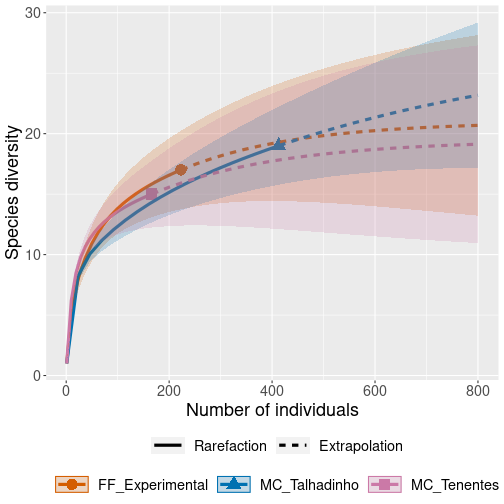
15 / 25
Exemplo de rarefação - Morcegos em 3 localidades
library(iNEXT)library(devtools)#devtools::install_github("paternogbc/ecodados")library(ecodados)dados_rarefacao <- rarefacao_morcegosresultados_morcegos <- iNEXT(dados_rarefacao, q = 0, datatype = "abundance", endpoint = 800)# q refere-se a família *Hill-numbers* (Hill 1973) onde 0 = riqueza de espécies, 1 = diversidade de Shannon, e 2 = diversidade de Simpson.# Veja mais detalhes sobre os números de Hill no Capítulo 7 onde tratamos de extrapolações.# datatype refere-se ao tipo de dados que você vai analisar (e.g. abudância, incidência).# endpoint refere-se ao valor de referência que você determina para a extrapolação.ggiNEXT(resultados_morcegos, type = 1)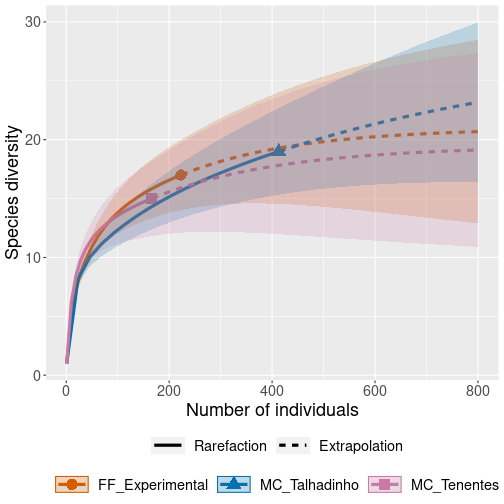
16 / 25
Índices de diversidade baseados em abundância
18 / 25
São uma tentativa de ponderar a riqueza de espécies pela abundância específica das espécies
19 / 25
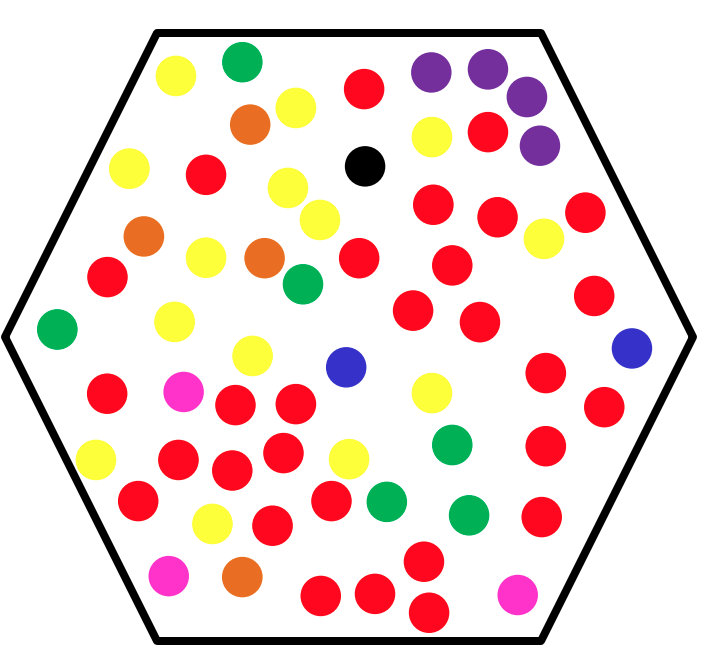
Quantas espécies existem nessa figura?

20 / 25
Quantas espécies existem nessa figura?

Riqueza = 5
20 / 25
Quantas espécies existem nessa figura?

21 / 25
Quantas espécies existem nessa figura?

Riqueza = 5
A riqueza desconsidera a abundância
21 / 25
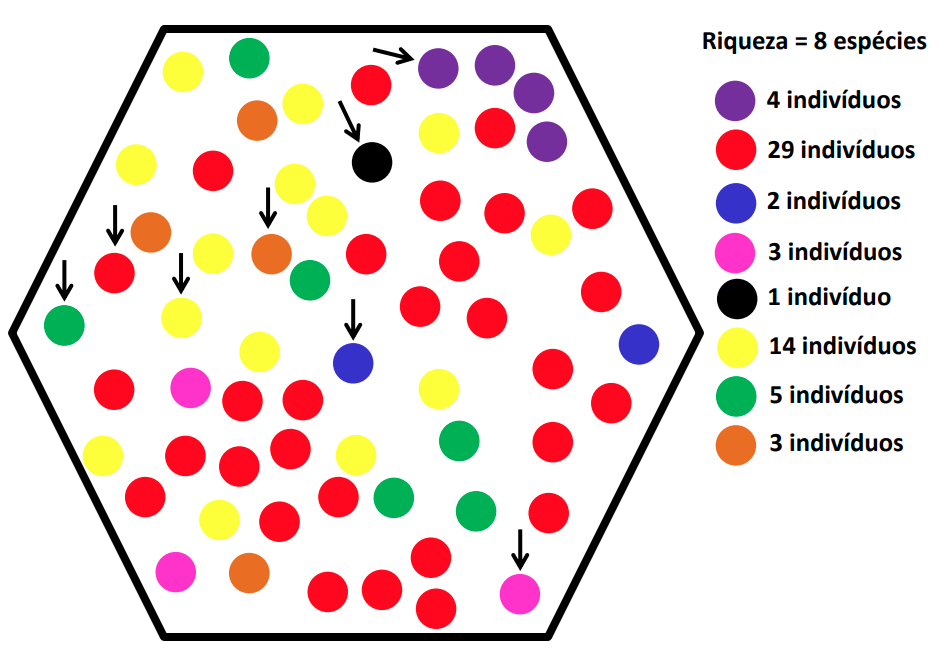
Quantas espécies existem nessa figura?

22 / 25
Quantas espécies existem nessa figura?

Riqueza = 5
A riqueza desconsidera a abundância
22 / 25
Riqueza + Abundância

Riqueza = 5
Índice de Shannon = 0,94
Riqueza = 5
Índice de Shannon = 1,61
23 / 25
Exemplos
panel-name[Código]
data(BCI)data("BCI.env")# ShannonH <- diversity(BCI)# Riquezarichness <- specnumber(BCI) # Equitatividade de Pielouevenness <- H/log(richness)# Create alpha diversity dataframe including environmental dataalpha <- cbind(shannon = H, richness = richness, pielou = evenness, BCI.env)alpha$site<-as.factor(seq(1:50))head(alpha)## shannon richness pielou UTM.EW UTM.NS elevation convex slope## 1 4.018412 93 0.8865579 625754 1011569 130.2525 -7.8725 6.694828## 2 3.848471 84 0.8685692 625754 1011669 136.8100 -10.7000 5.086842## 3 3.814060 90 0.8476046 625754 1011769 143.6775 -14.6675 3.104794## 4 3.976563 94 0.8752597 625754 1011869 147.0075 -16.7575 1.872813## 5 3.969940 101 0.8602030 625754 1011969 144.3850 -12.4850 5.118725## 6 3.776575 85 0.8500724 625854 1011569 136.8750 -9.6850 2.945532## aspectEW aspectNS site## 1 -0.89108252 -0.4538413 1## 2 -0.21903766 -0.9757164 2## 3 0.03051372 -0.9995343 3## 4 -0.86414183 -0.5032483 4## 5 -0.67148116 0.7410216 5## 6 -0.86532324 -0.5012142 6panel-name[Plots]
## Carregando pacotes exigidos: viridisLite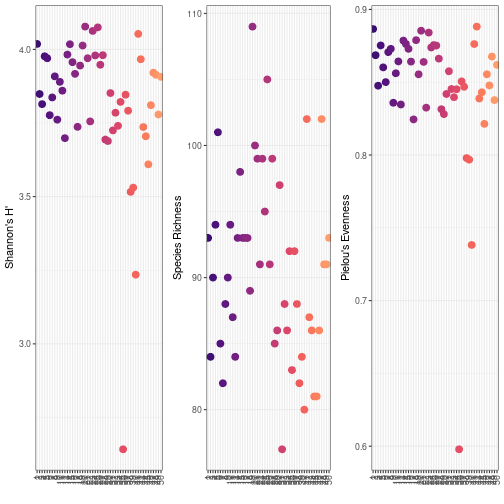
24 / 25
Vamos treinar
25 / 25